
ARTEMIS to nazwa nowej metody obliczeniowej, która może znacząco poszerzyć wiedzę na temat RNA, czyli kluczowej cząsteczki zaangażowanej w różne funkcje komórkowe. Algorytm opracowali naukowcy z Międzynarodowego Instytutu Biologii Molekularnej i Komórkowej w Warszawie.
RNA nie jest jedynie prostym łańcuchem nukleotydów, który przenosi informację genetyczną. Cząsteczki RNA pełnią także inne funkcje biologiczne, co często wiąże się z tworzeniem przez nie skomplikowanych struktur przestrzennych, podobnie jak w przypadku białek. Jednak porównanie złożonych kształtów struktur RNA okazuje się trudne, zwłaszcza gdy cząsteczki różnią się sekwencjami nukleotydów.
Ostatnie osiągnięcia biologii strukturalnej dają naukowcom możliwość bardziej efektywnego badania trójwymiarowych struktur RNA i lepszego rozumienia, w jaki sposób te struktury wpływają na funkcje RNA w organizmach. Jeżeli dwie cząsteczki RNA mają podobne struktury, można postawić i przetestować hipotezę, że posiadają one również podobne funkcje.
„W 2022 roku zespół zaprojektował nowy algorytm o nazwie ARTEM, oparty na założeniu, że w najlepszym możliwym nakładzie przestrzennym dwóch struktur RNA przynajmniej jeden nukleotyd z każdej struktury będzie niemal idealnie nałożony na swój odpowiednik” – przypomniał Międzynarodowy Instytut Biologii Molekularnej i Komórkowej (IIMCB) w Warszawie we wtorkowej informacji prasowej.
Opracowanie ARTEMIS było kolejnym krokiem, umożliwiającym porównywanie całych struktur cząsteczek RNA oraz ustalenie, które nukleotydy w jednej sekwencji odpowiadają nukleotydom w drugiej.
„Eugene F. Baulin wraz z zespołem przeprowadzili testy porównawcze i stwierdzili, że ARTEMIS działa bardzo dobrze – a dla wielu cząsteczek RNA nawet lepiej niż najlepsze narzędzia opracowane wcześniej przez innych badaczy” – czytamy w komunikacie.
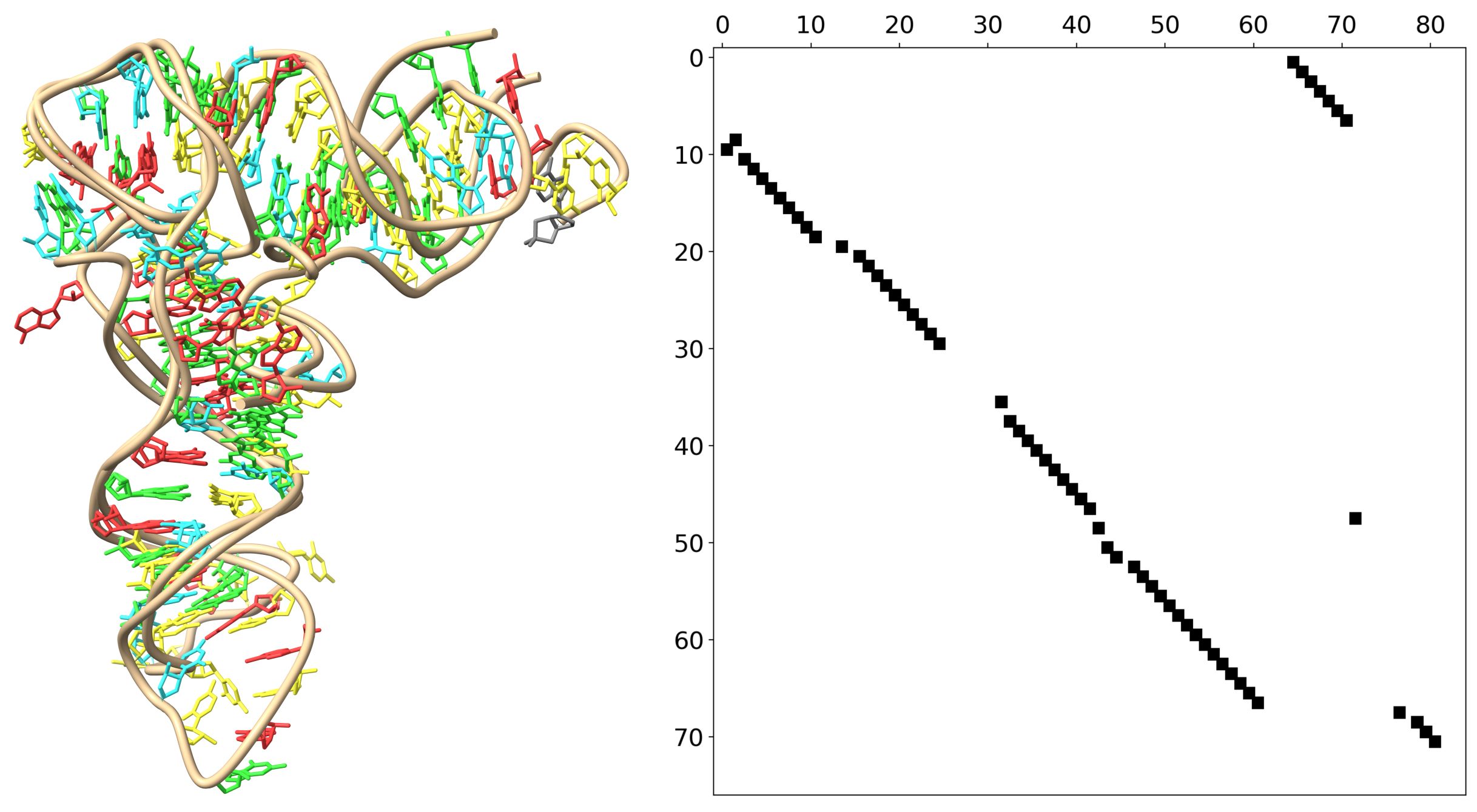
ARTEMIS, poprzez porównanie struktur cząsteczek RNA, jest w stanie z dużą dokładnością ustalić, które nukleotydy odpowiadają sobie nawzajem. Potwierdza to, że założenie badawcze dotyczące identyfikacji motywów strukturalnych poprzez nakładanie pojedynczych nukleotydów i weryfikację, jak mają się do siebie pozostałe nukleotydy, daje bardzo dobre efekty przy porównywaniu struktur przestrzennych całych cząsteczek RNA.
ARTEMIS został specjalnie zaprojektowany do analizy trójwymiarowych kształtów RNA, nawet w przypadkach, gdy sekwencje bardzo się różnią, włącznie ze zmianą kolejności nukleotydów. Obecne narzędzia często mają trudności z tak postawionym zadaniem. „W przeciwieństwie do nich, ARTEMIS działa bez ograniczeń dotyczących kolejności nukleotydów w porównywanych sekwencjach, co pozwala naukowcom na bardziej precyzyjne i efektywne porównanie struktur RNA” – stwierdzili badacze.
Twierdzą oni, że nowa metoda może zainteresować m.in. naukowców zajmujących się bioinformatyką strukturalną, analizujących bazy danych struktur, klasyfikujących znane struktury oraz przewidujących nieznane jeszcze struktury cząsteczek RNA. Jest to również wartościowe narzędzie dla biologów strukturalnych, którzy doświadczalnie określają nowe struktury kwasów nukleinowych, zarówno RNA, jak i DNA. Ci naukowcy mogą używać ARTEMIS do przeszukiwania baz danych strukturalnych w poszukiwaniu podobnych struktur lub motywów, które dostarczają wskazówek dotyczących funkcji biologicznej.
ARTEMIS może również przyczynić do lepszego projektowania nowych funkcjonalnych cząsteczek RNA i cząsteczek oddziałujących z RNA, mających zastosowanie w biotechnologii i medycynie. Jak zauważa Baulin, praktycznie każde wprowadzenie prawdziwie innowacyjnego wynalazku do codziennego zastosowania wywodziło się z wcześniejszych badań podstawowych – takich jak te, w ramach których powstała metoda ARTEMIS.
„Najbardziej ekscytuje mnie, jak jedno proste założenie dotyczące struktury RNA doprowadziło do opracowania potężnych narzędzi, które pozwalają nam dokonywać nowych odkryć” – powiedział Eugene F. Baulin. Jego zdaniem pokazuje to, że „znajomość danych może prowadzić do prostego rozwiązania, które przewyższa bardziej skomplikowane podejścia”.
„W związku z sukcesem algorytmu sztucznej inteligencji AlphaFold w przewidywaniu struktury białek, przewidywanie struktury RNA stało się teraz kolejnym świętym Graalem biologii obliczeniowej, a każde nowe narzędzie, takie jak ARTEMIS, przybliża nas nieco do tego rozwiązania” – podsumował naukowiec z Laboratorium Bioinformatyki i Inżynierii Białka IIMCB.(PAP)
Urszula Kaczorowska
uka/ bar/
Fundacja PAP zezwala na bezpłatny przedruk artykułów z Serwisu Nauka w Polsce pod warunkiem mailowego poinformowania nas raz w miesiącu o fakcie korzystania z serwisu oraz podania źródła artykułu. W portalach i serwisach internetowych prosimy o zamieszczenie podlinkowanego adresu: Źródło: naukawpolsce.pl, a w czasopismach adnotacji: Źródło: Serwis Nauka w Polsce - naukawpolsce.pl. Powyższe zezwolenie nie dotyczy: informacji z kategorii "Świat" oraz wszelkich fotografii i materiałów wideo.













