Twórcy nowych narzędzi do badania białek zostali laureatami Nagrody Nobla z chemii. David Baker, Demis Hassabis i John M. Jumper dali nauce metody obliczeniowe do projektowania i przewidywania trójwymiarowej struktury białek.
Naukowcy z USA i Wielkiej Brytanii: David Baker, Demis Hassabis i John M. Jumper zostali laureatami Nagrody Nobla z chemii za projektowanie i przewidywanie trójwymiarowej struktury białek - ogłosił w środę w Sztokholmie Komitet Noblowski.

"Jedno z odkryć, które zostaną docenione w tym roku, dotyczy budowy spektakularnych białek. Drugie dotyczy spełnienia 50-letniego marzenia: przewidywania struktur białek na podstawie ich sekwencji aminokwasowych. Oba te odkrycia otwierają ogromne możliwości" — powiedział Heiner Linke, przewodniczący Komitetu Noblowskiego w dziedzinie chemii.
Białka to związki organiczne o niezwykłych możliwościach - kontrolują i napędzają wszystkie reakcje chemiczne, które razem stanowią podstawę życia. Pełnią także funkcję hormonów, substancji sygnałowych, przeciwciał i budulca różnych tkanek.
Białka składają się zazwyczaj z 20 różnych aminokwasów, które można opisać jako elementy składowe życia. Wykorzystując informacje zapisane w DNA jako plan, aminokwasy są łączone ze sobą w naszych komórkach, tworząc długie łańcuchy. Łańcuchy te fałdują się w charakterystyczną i stabilną trójwymiarową strukturę, która jest decydująca dla funkcji białka. Aminokwasy mogą się łączyć w niekończenie różnorodnych kombinacjach: znanych jest około 200 milionów różnych białek – twardych, miękkich, elastycznych przezroczystych, świecących, bakteriobójczych, sygnalizacyjnych, kurczliwych... Od lat naukowcy próbowali przewidywać struktury białek na podstawie sekwencji aminokwasów, ale było to niezwykle trudne.
O tym, że białka są ważne dla procesów życiowych, chemicy wiedzą od XIX wieku, ale dopiero od ok. pół wieku narzędzia stały się dość precyzyjne, by białka można było badać bardziej szczegółowo. Naukowcy z Cambridge, John Kendrew i Max Perutz, dokonali przełomowego odkrycia, stosując z powodzeniem pod koniec dekady metodę zwaną krystalografią rentgenowską, by przedstawić pierwsze trójwymiarowe modele białek. W uznaniu tego odkrycia przyznano im Nobla w dziedzinie chemii w 1962 r.
Kolejnego wczesnego odkrycia dokonał Amerykanin Christian Anfinsen. Sprawił, że istniejące białko uległo rozłożeniu się, a następnie ponownie się złożyło. Ciekawą obserwacją było to, że cząsteczka białka za każdym razem przybierała dokładnie ten sam kształt. W 1961 r. doszedł do wniosku, że trójwymiarowa struktura białka jest zdeterminowana przez sekwencję aminokwasów. To doprowadziło do przyznania mu Nobla z chemii w 1972 r.
Logika Anfinsena zawiera jednak paradoks, na który zwrócił uwagę w 1969 r. inny Amerykanin, Cyrus Levinthal. Obliczył on, że nawet jeśli białko składa się tylko ze 100 aminokwasów, teoretycznie może przyjąć co najmniej 10 do potęgi 47 różnych trójwymiarowych struktur. Gdyby łańcuch aminokwasów fałdował się losowo, znalezienie prawidłowej struktury białka zajęłoby więcej czasu niż wiek wszechświata. W komórce zajmuje to zaledwie kilka milisekund. Jak to możliwe?
Odkrycie Anfinsena i paradoks Levinthala sugerowały, że fałdowanie białek jest z góry ustalonym procesem. I, co ważne, wszystkie informacje o tym, jak fałduje się białko, muszą być obecne w sekwencji aminokwasów.
Te spostrzeżenia doprowadziły do kolejnego wniosku: jeśli chemicy znają sekwencję aminokwasów białka, powinni też przewidzieć jego trójwymiarową strukturę. Gdyby to się udało, nie byłoby już potrzeby używać skomplikowanej i czasochłonnej krystalografii rentgenowskiej. Można by też wygenerować struktury wszystkich białek, dla których krystalografia rentgenowska nie miałaby zastosowania.
Aby zachęcić do szybszego rozwoju w tej dziedzinie, w 1994 roku naukowcy rozpoczęli projekt o nazwie Critical Assessment of Protein Structure Prediction (CASP), który przerodził się w konkurs. Co dwa lata badacze z całego świata otrzymywali dostęp do sekwencji aminokwasów w białkach, których struktury właśnie określono. Jednak struktury te były utrzymywane w tajemnicy przed uczestnikami. Wyzwaniem było przewidzenie struktur białek na podstawie znanych sekwencji aminokwasów.
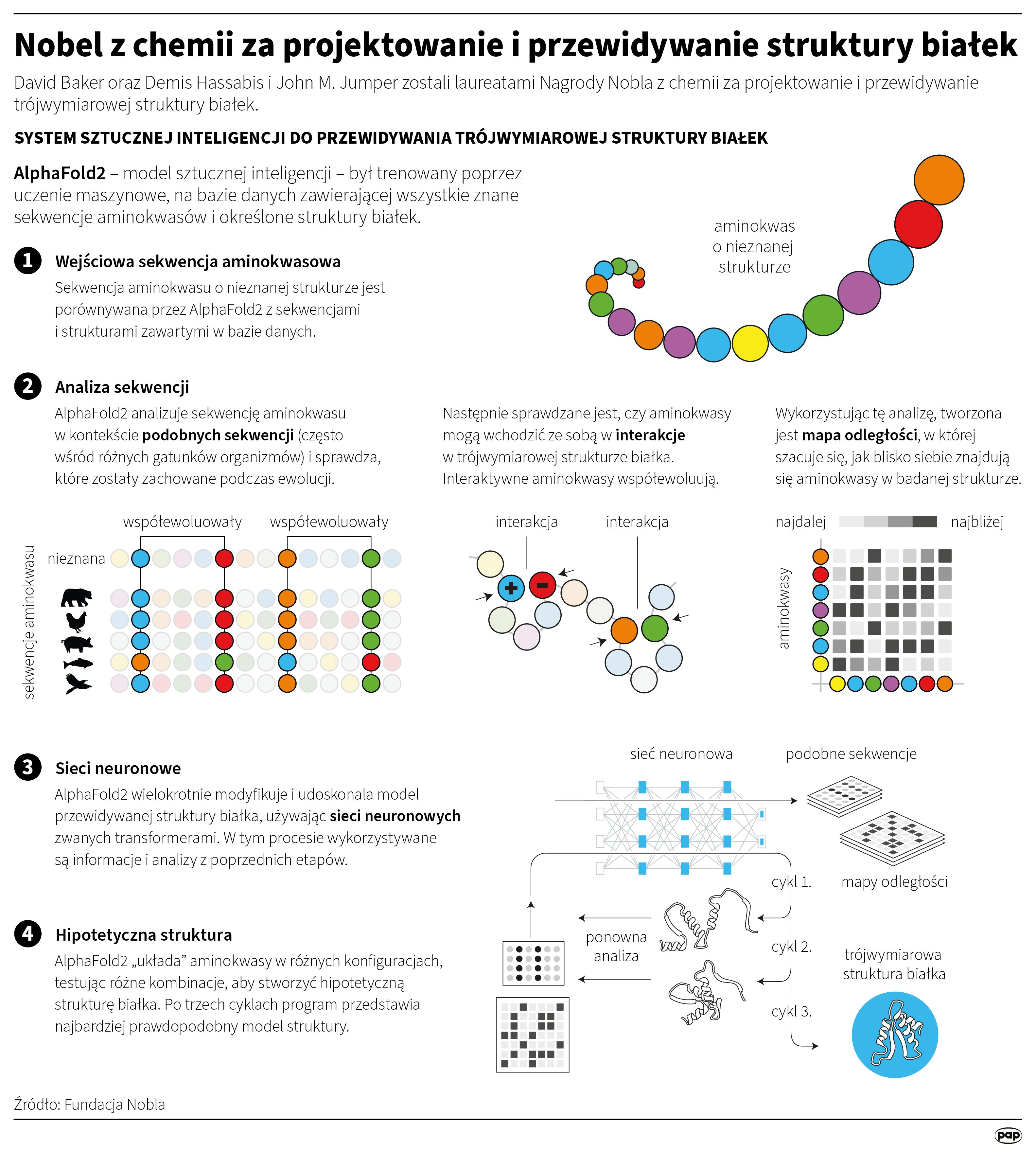
CASP przyciągnął wielu badaczy, ale przewidywanie okazało się trudne. Zgodność między prognozami, które badacze podali w konkursie - a rzeczywistymi strukturami, prawie wcale się nie poprawiła. Przełom nastąpił dopiero w 2018 r., gdy do gry wkroczył mistrz szachowy, ekspert w dziedzinie neuronauki i pionier sztucznej inteligencji: Demis Hassabis.
W poprzednich latach struktury białek, które naukowcy przewidywali dla CASP, osiągały dokładność wynoszącą co najwyżej 40 proc. Dzięki swojemu modelowi AI, AlphaFold, zespół Hassabisa osiągnął prawie 60 procent. Taki wynik zaskoczył, ale rozwiązanie nadal nie było wystarczająco dobre. Aby odnieść sukces, przewidywanie musiało mieć dokładność wynoszącą 90 procent w porównaniu do docelowej struktury.
W roku 2020 dzięki nowym pomysłom Johna Jumpera nastąpił przełom. Hassabis i Jumper zaprezentowali ulepszony model sztucznej inteligencji o nazwie AlphaFold2. Z jego pomocą byli w stanie przewidzieć strukturę praktycznie wszystkich znanych 200 milionów białek. Od czasu przełomu AlphaFold2 zostało wykorzystane przez ponad dwa miliony ludzi ze 190 krajów. Naukowcy mogą teraz np. lepiej zrozumieć oporność na antybiotyki i tworzyć obrazy enzymów, które mogą rozkładać plastik.
W 2003 roku Davidowi Bakerowi udało się wykorzystać aminokwasy do zaprojektowania nowego białka, które różniło się od wszystkich dotychczas znanych. Od tego czasu jego grupa badawcza tworzyła nowatorskie białka jedno po drugim, w tym takie, które można wykorzystać jako produkty farmaceutyczne, szczepionki, nanomateriały i mikroskopijne czujniki.
David Baker, urodzony 6 października 1952 w Seattle (USA), jest profesorem na University of Washington. Uważa się go za pioniera obliczeniowych metod projektowania (jak również przewidywania) trójwymiarowych struktur białek z wykorzystaniem sztucznej inteligencji. Jest założycielem wielu form biotechnologicznych. O tym, jak ważną rolę odegrały jego badania w rozwoju medycyny, świadczy fakt, że w 2024 r. magazyn "Time" uznał go za jedną ze stu najbardziej wpływowych osób w dziedzinie zdrowia.
"Jestem naprawdę podekscytowany w związku z tym, jak projektowanie białek czyni świat lepszym pod względem zdrowia, medycyny i również poza dziedziną technologii" - skomentował David Baker, gdy otrzymał telefoniczną informację o Nagrodzie Nobla podczas konferencji prasowej zorganizowanej przez Komitet Noblowski.
39-letni Brytyjczyk Demis Hassabis (ur. 27 lipca 1976) rozpoczynał karierę między innymi od projektowania i programowania gier komputerowych przy pomocy AI. Jest współzałożycielem m.in. brytyjsko-amerykańskiego laboratorium badawczego DeepMind (spółka zależna Google), zajmującego się sztuczną inteligencją. Doradza rządowi Wielkiej Brytanii w dziedzinie rozwoju technologii AI.
John Michael Jumper urodził się w 1981 w Little Rock w USA. Wraz z Hassabisem, w laboratorium DeepMind, stworzył system sztucznej inteligencji AlphaFold - model, który pozwala dokładnie przewidywać trójwymiarowe struktury białek na podstawie sekwencji ich aminokwasów. W 2021 r. naukowe czasopismo "Nature" zaliczyło go do dziesiątki najbardziej "znaczących" osób w dziedzinie nauki. Obecnie pracuje w DeepMind w Londynie.
Głównych autorów algorytmu AlphaFold szybko zaczęto wymieniać jako poważnych kandydatów do Nobla. Publikacja dotycząca ich pracy w "Nature" z 2020 r. cytowana była ponad 13 tys. razy. To ogromne osiągnięcie - spośród 61 mln publikacji naukowych tylko około 500 było cytowanych częściej niż 10 tys. razy.
Wszyscy trzej naukowcy byli wielokrotnie honorowani. Otrzymali m.in. The Wiley Prize w dziedzinie nauk biomedycznych, a także Breakthrough Prize w dziedzinie nauk o życiu: Baker w 2021 roku, Jumper z Hassabisem - w 2023 r. Ci ostatni dwaj są też laureatami Nagrody im. Alberta Laskera za wybitne osiągnięcia w badaniach medycznych.
Połowę Nagrody Nobla, wynoszącej 11 mln koron szwedzkich (4,2 mln zł), otrzyma Baker. Drugą połową podzielą się Hassabis z Jumperem.
Nauka w Polsce, Joanna Morga, Paweł Wernicki
jjj/ zan/ agt/
Fundacja PAP zezwala na bezpłatny przedruk artykułów z Serwisu Nauka w Polsce pod warunkiem mailowego poinformowania nas raz w miesiącu o fakcie korzystania z serwisu oraz podania źródła artykułu. W portalach i serwisach internetowych prosimy o zamieszczenie podlinkowanego adresu: Źródło: naukawpolsce.pl, a w czasopismach adnotacji: Źródło: Serwis Nauka w Polsce - naukawpolsce.pl. Powyższe zezwolenie nie dotyczy: informacji z kategorii "Świat" oraz wszelkich fotografii i materiałów wideo.